Wenn Wissenschaftler:innen vom genetischen Code sprechen, meinen sie meist den bekannten Prozess, bei dem DNA in Proteine übersetzt wird – die molekularen Maschinen des Lebens. Doch was, wenn das nur die halbe Geschichte ist?
Eine neue Studie des NanoPrecMed-Mitglieds Andrew Miller und seines Teams bringt eine gewagte Idee ins Spiel: Der genetische Code könnte nicht nur festlegen, welche Aminosäuren ein Protein bilden, sondern auch, wie sich diese Aminosäuren im Raum anordnen – also falten – und wie Proteine miteinander interagieren. Sie nennen dieses Prinzip den „universellen Proteom-Code“.
Vom Code zur Struktur: Warum Faltung entscheidend ist
Schon in der Schule lernen wir, dass jede Drei-Buchstaben-Kombination der DNA (ein Codon) für eine von 20 Aminosäuren codiert. Aneinandergereiht ergeben diese Aminosäuren die Grundstruktur eines Proteins. Doch Proteine funktionieren nicht als einfache Ketten – sie müssen sich in eine ganz bestimmte 3D-Form falten, um etwa als Enzyme, Antikörper oder Muskelbestandteile zu wirken. Falsch gefaltete Proteine können nicht nur nutzlos sein, sondern auch Krankheiten wie Alzheimer oder Parkinson verursachen.
Obwohl moderne KI-Modelle große Fortschritte bei der Vorhersage von Faltungsprozessen gemacht haben, bleiben sie oft eine Black Box: Sie zeigen meist, was passiert, aber nicht unbedingt warum. Genau hier setzt die neue Studie an.
Ein anderer Code im Code?
Die von Yazan Haddad und Andrew Miller an der Mendel-Universität in Tschechien geleitete Studie basiert auf einer Hypothese Millers, die bereits vor über 20 Jahren entstand: Im genetischen Code müsse eine zusätzliche Schicht an Information enthalten sein: ein „Proteom-Code“ oder eine „zweite genetische Codierung“, die Interaktionen zwischen Aminosäuren mitbestimmt.
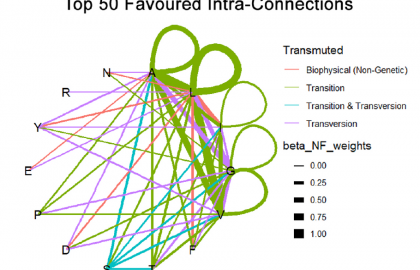
Auf Basis biophysikalischer und genetischer Prinzipien identifizierte das Team drei neue Modelle der Aminosäurepaarung (GU, Transmuted und Shift)1 die sich mit hoher statistischer Signifikanz in allen Hauptstrukturen von Proteinen nachweisen ließen. Diese Modelle bilden die Grundlage des „universellen Proteom-Codes“, der laut den Forschenden erklärt, warum bestimmte Aminosäuren einander räumlich anziehen oder meiden und so Proteinstruktur und -funktion steuern.
Ein universeller Code?
Ist das nun wirklich eine „zweite“ genetische Codierung? Die Forschenden sagen: Ja. Sie argumentieren, dass in der Redundanz und Struktur des bekannten genetischen Codes eine zusätzliche, logisch ableitbare Information enthalten ist – quasi eine unsichtbare Regieanweisung für die Proteinfaltung.
Wenn sich dieser Code bestätigt und etabliert, könnte er nicht nur helfen, Proteine gezielt zu entwerfen (etwa für Medikamente, Impfstoffe oder synthetische Materialien) sondern auch unser Verständnis genetischer Mutationen und ihrer Auswirkungen auf Krankheiten grundlegend verändern. Selbst die Lehre der Molekularbiologie müsste neu gedacht werden.
Was kommt als Nächstes?
Falls sich der Proteom-Code bewährt – und die Hinweise verdichten sich – könnte das weitreichende Folgen haben: von rationalen Vorhersagen proteinbasierter Strukturen bis hin zu neuen genetischen Therapien. Bemerkenswert ist, dass die Forschenden dabei nicht auf Big Data oder Machine Learning setzen. Stattdessen nutzen sie logische, genetische und biophysikalische Prinzipien zur Entwicklung erklärbarer Modelle.
Andrew Miller bringt es so auf den Punkt: „Es ist nicht anti-AI – es ist post-AI.“
Die Studie weist auf eine tiefere Logik in der Biologie hin, die Struktur und Funktion auf einer bisher unerkannten Ebene miteinander verknüpft. Denn: In der Natur ist selten etwas zufällig. Wenn es eine zweite Schicht genetischer Information gibt, dann vermutlich, weil sie gebraucht wird.